Bcl-2


Certaines informations figurant dans cet article ou cette section devraient être mieux reliées aux sources mentionnées dans les sections « Bibliographie », « Sources » ou « Liens externes » ().
Vous pouvez améliorer la vérifiabilité en associant ces informations à des références à l'aide d'appels de notes.
| BCL2 | |||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Identifiants | |||||||||||||||||||||||||||||||||||||||||||||||||||
| Aliases | BCL2 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| IDs externes | OMIM: 151430 MGI: 88138 HomoloGene: 527 GeneCards: BCL2 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Wikidata | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
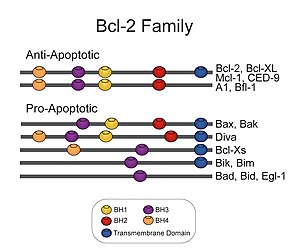
Bcl-2 est le prototype d'une famille de gènes qui peuvent être soit pro-apoptotiques – entre autres Bax, Bak, Bok, Bad, Bid et Bim – ou anti-apoptotiques – parmi lesquels Bcl-2, Bcl-xL, Bcl-w, Nr13 (ou Nrh - DIVA/BOO). À l'heure actuelle, il y a en tout 30 membres identifiés chez les eucaryotes supérieurs. La protéine Bcl-2 est une protéine de 239 acides aminés dont le gène est situé sur le chromosome 18 humain au locus q21.33. Elle est constituée de quatre domaines d’homologie qui sont aussi présents chez d’autres protéines de la même famille. Ce sont ces domaines, ainsi que son domaine trans-membranaire qui lui permettent d’avoir une action sur l’apoptose.
Origine du nom et lien avec le cancer
Bcl-2 doit son nom à l'anglais B-cell lymphoma 2 (lymphome à cellules B). Ce nom vient du fait que Bcl-2 fut la deuxième protéine à être découverte en lien avec le lymphome folliculaire humain à cellules B. Ce cancer est dû en grande partie à une translocation des chromosomes 14 et 18. Cette translocation fait en sorte de placer le gène de Bcl-2 à la suite du promoteur du gène des chaînes lourdes des immunoglobulines. Cela a pour effet de créer une surexpression de Bcl-2 et donc d’augmenter de façon substantielle la survie cellulaire des tissus exprimant normalement le gène des immunoglobulines, d’où le lymphome.
Dans divers cancers, on a retrouvé divers membres de la famille Bcl-2 qui avaient une expression altérée, ce qui implique que des troubles de l'apoptose peuvent être une cause de cancer. De même, sa présence est soupçonnée d’avoir un lien avec les résistances aux traitements anti-cancéreux conventionnels. Un exemple marquant est la surexpression de Bcl-2 dans un lymphome malin B particulier : le lymphome malin non Hodgkinien folliculaire. Elle est dans ce cas liée à la translocation t (14;18).
Bcl-2 est présente dans plusieurs tissus, tous caractérisés par un contrôle apoptotique de la survie cellulaire. Ces principaux tissus sont les tissus lymphoïdes, les épithéliums glandulaires, la peau, les intestins et les neurones. Dans ce tissu, Bcl-2 est principalement exprimé dans les cellules progénitrices ou effectrices, selon que le tissu est caractérisé par un haut taux de multiplication cellulaire ou par une longue survie cellulaire respectivement. De cette façon, ces cellules bénéficient d’une plus longue survie et peuvent ainsi accomplir leurs fonctions. Prenons par exemple les cellules mémoires du système immunitaire, sans cette longue vie, elles ne pourraient pas nous assurer une protection à long terme contre les infections.
Bcl-2, une protéine anti-apoptotique
La protéine Bcl-2 est capable d'inhiber l'apoptose. Localisée dans la membrane mitochondriale, elle empêche l'homodimérisation de Bax, de Bak et de certaines autres protéines pro-apoptotiques multi-domaines de la même famille. L'homodimère de Bax se fixe à la membrane externe mitochondriale. Cette fixation entraîne une perforation de la membrane mitochondriale et un relargage de cytochrome c dans le cytosol. Ceci entraîne une modification du potentiel membranaire mitochondrial ainsi qu'une perturbation de la chaîne respiratoire, mais ce n'est pas la cause de l'apoptose. En fait, c’est la présence de cytochrome c dans le cytosol qui cause cette dernière. Effectivement, en se liant à Apaf-1 et à la caspase 9, le cytochrome c crée un complexe nommé apoptosome. C’est ce complexe qui permet l'activation de caspases effectrices : caspase 3 et 7. C’est de cette façon que la chaîne des caspases est activée et entraîne la mort cellulaire. (Voir liens vers images pour des schémas)
Bcl-2 peut être inhibée par une protéine comme Bad. Bad est une protéine de la famille des Bcl-2 de type BH3 seulement, ce qui signifie qu'elle ne possède qu'un seul domaine d'homologie avec les autres protéines de cette famille, en l'occurrence, le domaine BH3. En conditions pro-apoptotiques, elle forme un lien avec Bcl-2 et l'empêche de se lier aux protéines pro-apoptotiques multi-domaines telles Bax et Bak. Dans ces conditions, Bcl-2 n'empêche plus l'homodimérisation de Bax ou de Bak. Ces dernières peuvent donc s'homodimériser pour former des pores. Le cytochrome C peut donc sortir de la mitochondrie et activer l'apoptose.
De plus, selon la voie qui active la cellule vers la mort cellulaire programmée, il est possible que l’inhibition par Bcl-2 soit contournée. Ainsi, si l’apoptose est activée par liaison du ligand de Fas avec son récepteur, il se produit une accumulation de caspase-3 dans le cytosol. Dans ces conditions, la caspase-3 est capable de cliver Bcl-2 dans sa boucle de l’extrémité NH2. Ce clivage se produit juste après l’acide aspartique en position 34 dans la séquence suivante ; Asp31-Ala32-Gly33-Asp34.
Ce clivage a pour effet, non seulement d’altérer les fonctions anti-apoptotiques de Bcl-2, mais aussi de lui conférer un pouvoir pro-apoptotique. Dans ces conditions, le processus de mort cellulaire est accéléré par la présence de Bcl-2 clivé. Les processus par lesquels Bcl-2 accélère l’apoptose à la suite de son clivage sont encore inconnus.
Cible thérapeutique
Plusieurs molécules inhibitrices du Bcl-2 ont été développées, avec une action pro-apoptose. Elles sont en test dans le traitement de certaines leucémies. Le navitoclax et le venetoclax sont en cours d'essai.
Quelques membres de la famille BCL-2
Notes et références
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Bcl-2 » (voir la liste des auteurs).
- ↑ a b et c GRCh38: Ensembl release 89: ENSG00000171791 - Ensembl, May 2017
- ↑ a b et c GRCm38: Ensembl release 89: ENSMUSG00000057329 - Ensembl, May 2017
- ↑ « Publications PubMed pour l'Homme », sur National Center for Biotechnology Information, U.S. National Library of Medicine
- ↑ « Publications PubMed pour la Souris », sur National Center for Biotechnology Information, U.S. National Library of Medicine
- ↑ (en) Debra T. Chao et Stanley J. Korsmeyer, « BCL-2 family: regulators of cell death », Annu Rev Immunol, vol. 16, , p. 395-419 (DOI 10.1146/annurev.immunol.16.1.395)
Voir aussi
Sur les autres projets Wikimedia :
- Bcl-2, sur Wikimedia Commons
Articles connexes
Bibliographie
- (en)YANG, Jie et al. « Prevention of Apoptosis by Bcl-2: Release of Cytochrome c from Mitochondria Blocked » Science, (page consultée le, 16 décembre 2007), [En ligne], http://www.sciencemag.org/cgi/content/full/275/5303/1129
- (en)KLUCK, Ruth M. et al. « The Release of Cytochrome c from Mitochondria: À Primary Site for Bcl-2 Regulation of Apoptosis » Science, (page consultée le 16 décembre 2007), [En ligne], http://www.sciencemag.org/cgi/content/full/275/5303/1132
- (en)HOCKENBERY, D M et al. « BCL2 protein is topographically restricted in tissues characterized by apoptotic cell death » PNAS, (page consultée le 16 décembre 2007), [En ligne], http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=52213
- (en)CHENG, Emily H.-Y. et al. « Conversion of Bcl-2 to a Bax-like Death Effector by Caspases » Science, (page consultée le 16 décembre 2007), [En ligne], http://www.sciencemag.org/cgi/content/full/278/5345/1966
Liens externes
- (en)The Bcl-2 Family Database
- Cours sur l'apoptose et autres processus cellulaires de la Faculté de médecine d'Angers
 Portail de la médecine
Portail de la médecine