| HIF1A |
|---|
 |
| Наявні структури |
|---|
| PDB | Пошук ортологів: PDBe RCSB |
|---|
| Список кодів PDB | | 1H2K, 1H2L, 1H2M, 1L3E, 1L8C, 1LM8, 1LQB, 2ILM, 3HQR, 3HQU, 4AJY, 4H6J | | |
| Ідентифікатори |
|---|
| Символи | HIF1A, HIF-1-alpha, HIF-1A, HIF-1alpha, HIF1, HIF1-ALPHA, MOP1, PASD8, bHLHe78, hypoxia inducible factor 1 alpha subunit, hypoxia inducible factor 1 subunit alpha, HIF-1α |
|---|
| Зовнішні ІД | OMIM: 603348 MGI: 106918 HomoloGene: 1171 GeneCards: HIF1A |
|---|
| Онтологія гена |
|---|
| Молекулярна функція | • protein dimerization activity
• GO:0001131, GO:0001151, GO:0001130, GO:0001204 DNA-binding transcription factor activity
• GO:0001077, GO:0001212, GO:0001213, GO:0001211, GO:0001205 DNA-binding transcription activator activity, RNA polymerase II-specific
• histone deacetylase binding
• transcription factor binding
• enzyme binding
• GO:0001948, GO:0016582 protein binding
• histone acetyltransferase binding
• protein kinase binding
• Hsp90 protein binding
• sequence-specific DNA binding
• DNA binding
• transcription factor activity, RNA polymerase II distal enhancer sequence-specific binding
• ubiquitin protein ligase binding
• protein heterodimerization activity
• E-box binding
• p53 binding
• protein domain specific binding
• GO:0001200, GO:0001133, GO:0001201 DNA-binding transcription factor activity, RNA polymerase II-specific
• RNA polymerase II transcription regulatory region sequence-specific DNA binding
|
|---|
| Клітинна компонента | • цитоплазма
• гіалоплазма
• клітинне ядро
• nuclear speck
• motile cilium
• transcription regulator complex
• RNA polymerase II transcription regulator complex
• axon cytoplasm
• нуклеоплазма
• ядерні тільця
• GO:0009327 protein-containing complex
|
|---|
| Біологічний процес | • GO:1904089 negative regulation of neuron apoptotic process
• B-1 B cell homeostasis
• regulation of transforming growth factor beta2 production
• muscle cell cellular homeostasis
• outflow tract morphogenesis
• negative regulation of ossification
• heart looping
• negative regulation of TOR signaling
• blood vessel development
• hemoglobin biosynthetic process
• Ангіогенез
• positive regulation of chemokine-mediated signaling pathway
• positive regulation of insulin secretion involved in cellular response to glucose stimulus
• positive regulation of hormone biosynthetic process
• negative regulation of mesenchymal cell apoptotic process
• regulation of aerobic respiration
• positive regulation of neuroblast proliferation
• dopaminergic neuron differentiation
• lactate metabolic process
• GO:0009373 regulation of transcription, DNA-templated
• blood vessel morphogenesis
• positive regulation of erythrocyte differentiation
• glucose homeostasis
• vascular endothelial growth factor production
• regulation of thymocyte apoptotic process
• positive regulation of epithelial cell migration
• negative regulation of thymocyte apoptotic process
• transcription, DNA-templated
• GO:0060469, GO:0009371 positive regulation of transcription, DNA-templated
• axonal transport of mitochondrion
• positive regulation of macroautophagy
• cartilage development
• positive regulation of nitric-oxide synthase activity
• regulation of transcription from RNA polymerase II promoter in response to hypoxia
• лактація
• negative regulation of oxidative stress-induced neuron intrinsic apoptotic signaling pathway
• positive regulation of pri-miRNA transcription by RNA polymerase II
• digestive tract morphogenesis
• диференціація клітин
• neural fold elevation formation
• positive regulation of autophagy
• retina vasculature development in camera-type eye
• collagen metabolic process
• embryonic placenta development
• negative regulation of apoptotic process
• positive regulation of angiogenesis
• regulation of glycolytic process
• Епітеліально-мезенхімальний перехід
• cerebral cortex development
• регуляція експресії генів
• positive regulation of chemokine production
• intestinal epithelial cell maturation
• GO:0048552 regulation of catalytic activity
• positive regulation of autophagy of mitochondrion
• cardiac ventricle morphogenesis
• response to muscle activity
• epithelial cell differentiation involved in mammary gland alveolus development
• visual learning
• positive regulation of vascular endothelial growth factor receptor signaling pathway
• negative regulation of bone mineralization
• negative regulation of growth
• response to hypoxia
• positive regulation of endothelial cell proliferation
• regulation of transcription from RNA polymerase II promoter in response to oxidative stress
• iris morphogenesis
• mRNA transcription by RNA polymerase II
• hypoxia-inducible factor-1alpha signaling pathway
• vasculature development
• regulation of cell population proliferation
• neural crest cell migration
• embryonic hemopoiesis
• connective tissue replacement involved in inflammatory response wound healing
• positive regulation of transcription from RNA polymerase II promoter in response to hypoxia
• negative regulation of reactive oxygen species metabolic process
• positive regulation of vascular endothelial growth factor production
• elastin metabolic process
• positive regulation of glycolytic process
• oxygen homeostasis
• GO:0072468 сигнальна трансдукція
• GO:0003257, GO:0010735, GO:1901228, GO:1900622, GO:1904488 positive regulation of transcription by RNA polymerase II
• cellular iron ion homeostasis
• camera-type eye morphogenesis
• cellular response to hypoxia
• cellular response to interleukin-1
• transcription by RNA polymerase II
• protein deubiquitination
• посттрансляційна модифікація
• response to iron ion
• negative regulation of gene expression
• positive regulation of blood vessel endothelial cell migration
• GO:1901313 positive regulation of gene expression
• cytokine-mediated signaling pathway
• GO:0044324, GO:0003256, GO:1901213, GO:0046019, GO:0046020, GO:1900094, GO:0061216, GO:0060994, GO:1902064, GO:0003258, GO:0072212 regulation of transcription by RNA polymerase II
• Убіквітин-залежний протеоліз
|
|---|
| Джерела:Amigo / QuickGO | |
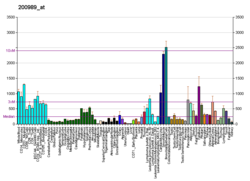
| Шаблон експресії |
|---|
 |
| Більше даних |
| Ортологи |
|---|
| Види | Людина | Миша |
|---|
| Entrez | | |
|---|
| Ensembl | | |
|---|
| UniProt | | |
|---|
| RefSeq (мРНК) | | NM_181054
NM_001243084
NM_001530 |
| | NM_010431
NM_001313919
NM_001313920 |
|
|---|
| RefSeq (білок) | | NP_001230013
NP_001521
NP_851397
NP_001521.1 |
| | NP_001300848
NP_001300849
NP_034561 |
|
|---|
| Локус (UCSC) | Хр. 14: 61.7 – 61.75 Mb | Хр. 12: 73.95 – 73.99 Mb |
|---|
| PubMed search | [1] | [2] |
|---|
| Вікідані |
| Див./Ред. для людей | Див./Ред. для мишей |
|












