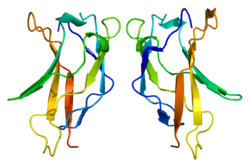| Наявні структури |
|---|
| PDB | Пошук ортологів: PDBe RCSB |
|---|
| Список кодів PDB | | 4KV4, 1NFI, 2LSP, 2O61, 3GUT, 3QXY, 3RC0, 4KV1, 2N22 | | |
| Онтологія гена |
|---|
| Молекулярна функція | • protein N-terminus binding
• ankyrin repeat binding
• GO:0001077, GO:0001212, GO:0001213, GO:0001211, GO:0001205 DNA-binding transcription activator activity, RNA polymerase II-specific
• histone deacetylase binding
• enzyme binding
• phosphate ion binding
• GO:0000980 RNA polymerase II cis-regulatory region sequence-specific DNA binding
• protein homodimerization activity
• chromatin binding
• NF-kappaB binding
• GO:0001948, GO:0016582 protein binding
• protein kinase binding
• DNA binding
• sequence-specific DNA binding
• actinin binding
• protein heterodimerization activity
• transcription factor activity, RNA polymerase II distal enhancer sequence-specific binding
• chromatin DNA binding
• ubiquitin protein ligase binding
• GO:0000983 RNA polymerase II general transcription initiation factor activity
• RNA polymerase II transcription regulatory region sequence-specific DNA binding
• GO:0001131, GO:0001151, GO:0001130, GO:0001204 DNA-binding transcription factor activity
• GO:0001078, GO:0001214, GO:0001206 DNA-binding transcription repressor activity, RNA polymerase II-specific
• identical protein binding
• GO:0001200, GO:0001133, GO:0001201 DNA-binding transcription factor activity, RNA polymerase II-specific
• transcription factor binding
• GO:0032403 protein-containing complex binding
|
|---|
| Клітинна компонента | • цитоплазма
• гіалоплазма
• I-kappaB/NF-kappaB complex
• transcription regulator complex
• NF-kappaB complex
• нуклеоплазма
• клітинне ядро
• ядерце
• NF-kappaB p50/p65 complex
• GO:0009327 protein-containing complex
• синапс
• glutamatergic synapse
|
|---|
| Біологічний процес | • response to amino acid
• response to interleukin-1
• cellular response to interleukin-6
• response to progesterone
• response to organic substance
• Fc-epsilon receptor signaling pathway
• GO:1903105 negative regulation of insulin receptor signaling pathway
• defense response to virus
• positive regulation of chondrocyte differentiation
• animal organ morphogenesis
• response to muscle stretch
• response to cytokine
• GO:1903363 negative regulation of protein catabolic process
• cytokine-mediated signaling pathway
• negative regulation of extrinsic apoptotic signaling pathway
• response to mechanical stimulus
• response to muramyl dipeptide
• stimulatory C-type lectin receptor signaling pathway
• transcription, DNA-templated
• response to insulin
• negative regulation of NIK/NF-kappaB signaling
• nucleotide-binding oligomerization domain containing 2 signaling pathway
• membrane protein intracellular domain proteolysis
• inflammatory response
• GO:0022415 viral process
• response to cobalamin
• hair follicle development
• positive regulation of miRNA metabolic process
• acetaldehyde metabolic process
• cellular response to hepatocyte growth factor stimulus
• cellular response to nicotine
• negative regulation of apoptotic process
• cellular defense response
• response to morphine
• response to lipopolysaccharide
• cellular response to interleukin-1
• response to inorganic substance
• regulation of inflammatory response
• response to cAMP
• GO:0045996 negative regulation of transcription, DNA-templated
• response to hydrogen peroxide
• positive regulation of Schwann cell differentiation
• defense response
• response to bacterium
• GO:1904578 response to organic cyclic compound
• GO:0010260 старіння людини
• positive regulation of cell population proliferation
• positive regulation of I-kappaB kinase/NF-kappaB signaling
• liver development
• response to UV-B
• T cell receptor signaling pathway
• cellular response to lipopolysaccharide
• positive regulation of type I interferon production
• GO:0003257, GO:0010735, GO:1901228, GO:1900622, GO:1904488 positive regulation of transcription by RNA polymerase II
• cellular response to hydrogen peroxide
• GO:0009373 regulation of transcription, DNA-templated
• tumor necrosis factor-mediated signaling pathway
• GO:0060469, GO:0009371 positive regulation of transcription, DNA-templated
• positive regulation of T cell receptor signaling pathway
• positive regulation of NF-kappaB transcription factor activity
• cellular response to peptidoglycan
• cellular response to tumor necrosis factor
• regulation of NIK/NF-kappaB signaling
• positive regulation of NIK/NF-kappaB signaling
• GO:0007329 positive regulation of transcription from RNA polymerase II promoter involved in cellular response to chemical stimulus
• positive regulation of pri-miRNA transcription by RNA polymerase II
• regulation of DNA-templated transcription in response to stress
• cellular response to lipoteichoic acid
• GO:1901227 negative regulation of transcription by RNA polymerase II
• negative regulation of protein sumoylation
• pri-miRNA transcription by RNA polymerase II
• cellular response to angiotensin
• interleukin-1-mediated signaling pathway
• cellular response to vascular endothelial growth factor stimulus
• negative regulation of pri-miRNA transcription by RNA polymerase II
• postsynapse to nucleus signaling pathway
• GO:0031497, GO:0006336, GO:0034724, GO:0001301, GO:0007580, GO:0034652, GO:0010847 chromatin organization
• I-kappaB kinase/NF-kappaB signaling
• NIK/NF-kappaB signaling
• positive regulation of leukocyte adhesion to vascular endothelial cell
|
|---|
| Джерела:Amigo / QuickGO | |